RNA-sekventering (2009ang 2009) erstatter hurtigt genekspressionsmikroarrays i mange laboratorier. RNA-se.lader dig kvantificere, opdage og profilere RNA ‘ er. Til denne teknik konverteres mRNA (og andre RNA ‘ er) først til cDNA. CDNA bruges derefter som input til en næste generations sekventering bibliotek forberedelse. I denne artikel vil jeg give en kort gennemgang af RNA-se.og introducere de vigtigste metoder, der bruges i dag.
Hvorfor er RNA-se? “bedre” end mikroarrays?,
Der er flere fordele RNA-se.har over microarrays:
med RNA-se. kan du afhøre mere end blot differential genekspression. Selvom der er mikroarrays tilgængelige til e .on-niveau og microRNA-analyse, er de fleste brugere stadig interesseret i grundlæggende, sandsynligvis 3′ partisk, differentieret genekspression. Med RNA-seq du kan se på kodende og ikke-kodende RNA, ved splejsning og allel-specifikke udtryk, og muligvis snart på full-length cDNA sekvenser, hvilket eliminerer behovet for at udlede eller samle isoformer.,
mikroarrays er også partiske, da vi skal beslutte, hvilket indhold der skal placeres på arrayet. Da RNA-se.ikke bruger prober eller primere, lider dataene af meget lavere forstyrrelser (selvom jeg ikke mener at sige RNA-se. har ingen).RNA-se.leverer digitale data i form af justerede læsetællinger, hvilket resulterer i et meget bredt dynamisk område, hvilket forbedrer følsomheden af detektion for sjældne transkripter.
det er også meget omkostningseffektivt for mikroarrays, da i dag kan mellem 6-30 prøver multiple .eres i en enkelt Illumina-sekventeringsbane.,
endelig kan du genanalysere et RNA-se. – datasæt, efterhånden som flere oplysninger om transkriptomet bliver tilgængelige. Hvis et papir er offentliggjort viser en interessant splice-variant i et lignende system til den, du arbejder på, så er du måske ønsker at gå tilbage og se på, at splejsning i dine prøver; og du ville allerede have data til at gøre det.
Hvordan virker RNA-se??
Der er mange metoder til udførelse af et RNA-se. – eksperiment. Faktisk udvikler teknikkerne sig så hurtigt, at det kan være svært at afgøre, hvilken man skal bruge., Et grundlæggende valg er mellem 1) tilfældig primet cDNA-syntese fra dobbeltstrenget cDNA eller 2) RNA-ligeringsmetoder (gennemgået og sammenlignet i Levin 2010). De fleste mennesker bruger den første metode og skal derefter foretage et yderligere valg mellem en strengspecifik protokol og en, der ikke er. Den metode, der bruges mest i mit laboratorium, er Illuminas Truse.RNA-se., som er en tilfældig primet cDNA-syntese, ikke-streng-specifik protokol.
Når du har et sekventeringsbibliotek, sekventeres det til en bestemt dybde, hvilket afhænger af, hvad du vil gøre med dataene., Disse læsninger er justeret til genomet eller transkriptomet og tælles for at bestemme differentiel genekspression eller yderligere analyseret for at bestemme splejsning og isoformekspression. De fleste mennesker er sekventering RNA ved hjælp af parret-end 50-100bp metoder. Undtagelsen er microRNA sekventering, da dette kun kræver single-end 36bp sekventering i de fleste tilfælde.
vores RNA-se.metode
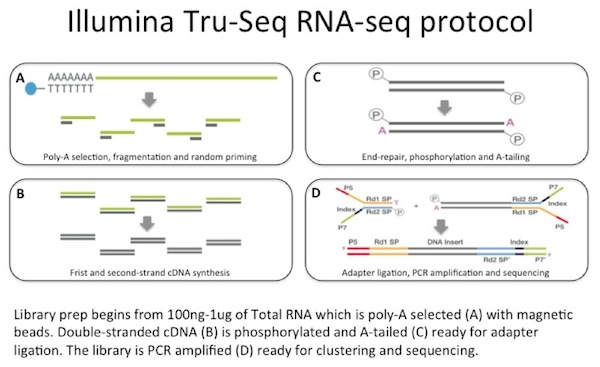
Vi bruger mellem 100 ng til 1 µg af total RNA som input til en mRNA capture med oligo-dt coatede magnetiske perler. MRNA er fragmenteret, og derefter udføres en tilfældig primet cDNA-syntese., Den resulterende dobbeltstrenget cDNA bruges som input til en standard Illumina bibliotek prep som inkluderer end-reparation, adapter ligering og PCR forstærkning til at give dig et bibliotek klar til sekventering.
hvorfor gider med Strenginformation?
der har været en masse diskussion om anti-sense transkription og dens biologiske relevans. Hvis du er interesseret i simpel differentiel genekspression, vil strenginformation ikke tilføje meget til dit eksperiment, men vil gøre din protokol mere kompleks., Når det er sagt, kan du udføre den mest anvendte metode uden for meget ekstra indsats. For at gøre dette skal du bruge uracil til inkorporering i stedet for thymin under 2.streng cDNA-syntese. Følg Illumina library prep som normalt, men efter adapterligation og før PCR-forstærkning tilsættes uracil-DNA glycosylase for at nedbryde 2ndstranden. Dette resulterer i, at alle læsninger starter i samme retning, så du kan bestemme, hvilken streng der blev transkriberet i din prøve.
Hvad kan du faktisk gøre med RNA-se??,
RNA-se.er et kraftfuldt og alsidigt værktøj udgivet bredt i de sidste par år. Jeg har valgt et par af mine favoritter (nogle fra arbejde udført i kernefaciliteten, jeg administrerer) for at illustrere, hvad du kan gøre med RNA-sekventering.
- Jabbari, et al. brugte RNA-se.til at undersøge psoriasis og finde nye gener til funktionel analyse. De sammenlignede deres RNA-se. – data med offentliggjorte array-undersøgelser og fandt 1700 nye kandidater. Disse blev valideret af psoripcr, og sammenligning med funktionelle databaser for psoriasis understøttede deres rolle i patogenese.
- Kutter, et al., brugt RNA-se.i en undersøgelse, der ser på bevarelsen af RNA-Polymerase III-binding hos pattedyr for at validere ekspression af gener besat af Pol III som analyseret af ChIP-se..
- Mercer, et al. kombineret RNA-se.og microarray-baseret capture til at identificere og karakterisere sjældne transkripter, som normalt ikke kan påvises. De målrettede udskrifter steg i rækkefølge læse overflod fra 0.21% pre-capture til 80% post capture., De fandt mere end 200 tidligere uden markeringer isoformer for næsten 50 protein-kodende loci, herunder en ny alternativ isoform af TP53, som er et meget godt karakteriseret gen. Dette antyder, at der stadig er meget kompleksitet i genomet og transkriptomet, der skal løses.
Sammenfattende er RNA-se.stadig et udviklende værktøj, men foretrækkes i de fleste tilfælde frem for mikroarrays. Det er mere følsomt, mere robust og kan være mere omkostningseffektivt. Hvilke RNA-se?projekter planlægger du nu til dit projekt?,transkriptionel profilering af Psoriasis ved anvendelse af RNA-se.afslører tidligere uidentificerede differentielt udtrykte gener. Journal of Investigative Dermatology 2011.Pol III-binding i seks pattedyr viser bevarelse blandt aminosyreisotyper på trods af divergens mellem tRNA-gener. Nature Genetics 2011.
Levin et al: omfattende sammenlignende analyse af strengspecifikke RNA-sekventeringsmetoder. Natur Metoder 2010.
Mercer et al: målrettet RNA-sekventering afslører den dybe kompleksitet af det humane transkriptom. Nature Biotechnology 2012.,RNAANG et al: RNA-se.: et revolutionerende værktøj til transkriptomik. Nat Rev Genet 2009.
oprindeligt offentliggjort den 25.maj 2012. Opdateret og revideret den 16. August 2015.

har dette hjulpet dig? Så kan du dele med dit netværk.