(Wang 2009) está reemplazando rápidamente los microarrays de expresión génica en muchos laboratorios. RNA-seq le permite cuantificar, descubrir y perfilar ARN. Para esta técnica, el ARNm (y otros ARN) se convierten primero en ADNc. El cDNA se utiliza entonces como la entrada para una preparación de biblioteca de secuenciación de próxima generación. En este artículo, voy a dar una breve revisión de RNA-seq e introducir los principales métodos que se utilizan hoy en día.
¿por qué es RNA-Seq «mejor» que los Microarrays?,
hay varias ventajas que tiene RNA-seq sobre los microarrays:
con RNA-seq puede interrogar más que solo la expresión génica diferencial. Aunque hay microarrays disponibles para el análisis del nivel del exón y del microRNA, la mayoría de los usuarios todavía están interesados en la expresión génica básica, probablemente 3 ‘ sesgada, diferencial. Con RNA-seq se puede observar ARN codificante y no codificante, empalme y expresión específica de alelos, y posiblemente pronto secuencias completas de ADNc, eliminando la necesidad de inferir o ensamblar isoformas.,
los Microarrays también están sesgados, ya que tenemos que decidir qué contenido colocar en el array. Dado que RNA-seq no utiliza sondas o cebadores, los datos sufren de sesgos mucho más bajos (aunque no quiero decir que RNA-seq no tiene ninguno).
RNA-seq proporciona datos digitales en forma de recuentos de lectura alineados, lo que resulta en un rango dinámico muy amplio, mejorando la sensibilidad de detección de transcripciones raras.
también es muy rentable para microarrays, ya que hoy en día, entre 6-30 muestras se pueden multiplexar en un solo carril de secuenciación Illumina.,
Por último, puede volver a analizar un conjunto de datos RNA-seq a medida que se disponga de más información sobre el transcriptoma. Si se publica un documento que muestra una variante de empalme interesante en un sistema similar al que trabaja, entonces es posible que desee volver y mirar ese empalme en sus muestras; y ya tendría los datos para hacerlo.
¿cómo actúa RNA-Seq?
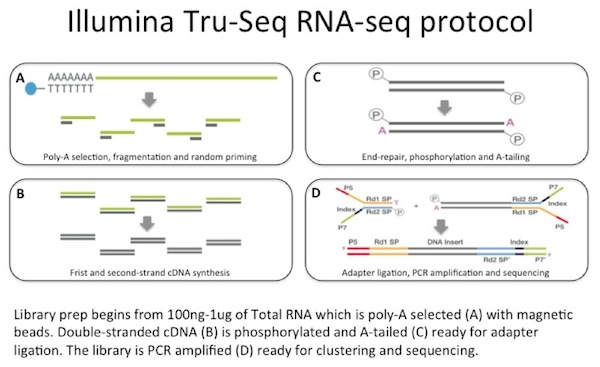
hay muchos métodos para realizar un experimento de ARN-seq. De hecho, las técnicas están evolucionando tan rápidamente que puede ser difícil decidir cuál usar., Una opción básica es entre 1) síntesis de ADNc de cebado aleatorio a partir de ADNc de doble cadena o 2) métodos de ligadura de ARN (revisado y comparado en Levin 2010). La mayoría de las personas usan el primer método y luego necesitan hacer una elección adicional entre un protocolo específico de la cadena y uno que no lo es. El método más utilizado en mi laboratorio es el TruSeq RNA-seq de Illumina, que es un protocolo de síntesis de ADNc de cebado Aleatorio no específico de hebras.
una vez que tenga una biblioteca de secuenciación, se secuenciará a una profundidad especificada, que depende de lo que desee hacer con los datos., Estas lecturas están alineadas con el genoma o transcriptoma y se cuentan para determinar la expresión génica diferencial o se analizan más a fondo para determinar el empalme y la expresión isoforma. La mayoría de las personas están secuenciando el ARN utilizando métodos de 50-100bp de extremo emparejado. La excepción es la secuenciación de microRNA, ya que solo requiere secuenciación de 36bp de un solo extremo en la mayoría de los casos.
nuestro método RNA-Seq
utilizamos entre 100 ng y 1 µg de ARN total como entrada para una captura de ARNm con perlas magnéticas recubiertas de oligo-dT. El ARNm se fragmenta, y luego se realiza una síntesis de ADNc al azar., El cDNA de doble hebra resultante se utiliza como entrada para una preparación de biblioteca Illumina estándar que incluye reparación final, ligadura de adaptador y amplificación de PCR para darle una biblioteca lista para secuenciar.
¿por qué molestarse con la información de Strand?
ha habido mucha discusión sobre la transcripción anti-sentido y su relevancia biológica. Si está interesado en la expresión génica diferencial simple, entonces la información de la cadena no agregará mucho a su experimento, pero hará que su protocolo sea más complejo., Dicho esto, puede realizar el método más ampliamente adoptado sin demasiado esfuerzo adicional. Para hacer esto, durante la síntesis de cDNA de 2da hebra, use uracilo para incorporación en lugar de timina. Siga la preparación de la Biblioteca Illumina como es normal, pero después de la ligadura del adaptador y antes de la amplificación de PCR agregue uracilo-ADN glicosilasa para degradar la 2a marca. Esto da como resultado que todas las lecturas comiencen en la misma orientación para que pueda determinar qué cadena se estaba transcribiendo en su muestra.
¿qué se puede hacer realmente con RNA-Seq?,
RNA-seq es una herramienta potente y versátil publicada ampliamente en los últimos años. He elegido un par de mis favoritos (algunos de trabajo realizado en la instalación central que administro) para ilustrar lo que puede hacer con la secuenciación de ARN.
- Jabbari, et al. se utilizó ARN-seq para investigar la psoriasis y encontrar nuevos genes para el análisis funcional. Compararon sus datos de RNA-seq con estudios publicados y encontraron 1700 nuevos candidatos. Estos fueron validados por qPCR, y la comparación con las bases de datos funcionales para la psoriasis apoyó su papel en la patogénesis.
- Kutter, et al., se utilizó ARN-seq en un estudio que analiza la conservación de la Unión de ARN polimerasa III en mamíferos para validar la expresión de genes ocupados por Pol III como ensayado por ChIP-seq.
- Mercer, et al. captura combinada de ARN-seq y microarray para identificar y caracterizar transcripciones raras, que normalmente son indetectables. Las transcripciones objetivo aumentaron en abundancia de lectura de secuencia de 0.21% antes de la captura a 80% después de la captura., Encontraron más de 200 isoformas no analizadas previamente para casi 50 loci codificadores de proteínas, incluida una nueva isoforma alternativa de TP53, que es un gen muy bien caracterizado. Esto sugiere que todavía hay mucha complejidad en el genoma y el transcriptoma por resolver.
En resumen, el RNA-seq sigue siendo una herramienta en evolución, pero es preferible en la mayoría de los casos a los microarrays. Es más sensible, más robusto y puede ser más rentable. ¿Qué proyectos de RNA-seq están planeando para su proyecto?,
Jabbari et al: el perfil transcripcional de la Psoriasis utilizando ARN-seq revela Genes expresados diferencialmente previamente no identificados. Journal of Investigative Dermatology 2011.
Kutter et al: La Unión de Pol III en seis mamíferos muestra conservación entre los isotipos de aminoácidos a pesar de la divergencia entre los genes del ARNt. Nature Genetics 2011.
Levin et al: Comprehensive comparative analysis of strand-specific RNA sequencing methods. Nature Methods 2010.
Mercer et al: la secuenciación de ARN dirigido revela la profunda complejidad del transcriptoma humano. Nature Biotechnology 2012.,
Wang et al: RNA-Seq: una herramienta revolucionaria para la transcriptómica. Nat Rev Genet 2009.
publicado originalmente el 25 de mayo de 2012. Actualizado y revisado el 16 de agosto de 2015.

Tiene esto ha ayudado? Entonces por favor comparta con su red.