le séquençage de L’ARN (Wang 2009) remplace rapidement les puces à expression génique dans de nombreux laboratoires. RNA-seq vous permet de quantifier, de découvrir et de profiler les ARN. Pour cette technique, l’ARNm (et d’autres ARN) sont d’abord convertis en ADNc. L’ADNc est ensuite utilisé comme entrée pour une préparation de bibliothèque de séquençage de nouvelle génération. Dans cet article, je vais donner un bref aperçu de L’ARN-seq et présenter les principales méthodes utilisées aujourd’hui.
pourquoi RNA-Seq est-il” meilleur » que les puces à puces?,
L’ARN-seq présente plusieurs avantages par rapport aux puces à puces:
avec L’ARN-seq, vous pouvez interroger plus que l’expression différentielle des gènes. Bien qu’il existe des puces disponibles pour l’analyse au niveau des exons et des microARN, la plupart des utilisateurs sont toujours intéressés par l’expression génique différentielle de base, probablement biaisée de 3’. Avec RNA-seq, vous pouvez examiner les ARN codants et non codants, l’épissage et l’expression spécifique des allèles, et peut-être bientôt les séquences d’ADNc complètes, éliminant ainsi le besoin de déduire ou d’assembler des isoformes.,
Les puces sont également biaisées, car nous devons décider quel contenu placer sur le tableau. Étant donné que RNA-seq n’utilise pas de sondes ou d’amorces, les données souffrent de biais beaucoup plus faibles (bien que je ne veuille pas dire que RNA-seq n’en a pas).
RNA-seq fournit des données numériques sous la forme de comptes de lecture alignés, ce qui donne une plage dynamique très large, améliorant la sensibilité de détection pour les transcriptions rares.
Il est également très compétitif sur le plan des coûts pour les puces, car aujourd’hui, entre 6 et 30 échantillons peuvent être multiplexés dans une seule voie de séquençage Illumina.,
enfin, vous pouvez réanalyser un ensemble de données ARN-seq à mesure que plus d’informations sur le transcriptome deviennent disponibles. Si un article est publié montrant une variante d’épissure intéressante dans un système similaire à celui sur lequel vous travaillez, vous voudrez peut-être revenir en arrière et regarder cette épissure dans vos échantillons; et vous auriez déjà les données pour le faire.
Comment fonctionne L’ARN-Seq?
Il existe de nombreuses méthodes pour effectuer une expérience RNA-seq. En fait, les techniques évoluent si rapidement qu’il peut être difficile de décider lequel utiliser., Un choix de base est entre 1) la synthèse d’ADNc à amorçage aléatoire à partir d’ADNc double brin ou 2) les méthodes de ligature de l’ARN (examinées et comparées dans Levin 2010). La plupart des gens utilisent la première méthode et doivent ensuite faire un choix supplémentaire entre un protocole spécifique au volet et un autre qui ne l’est pas. La méthode la plus utilisée dans mon laboratoire est le TruSeq RNA-seq D’Illumina, qui est un protocole de synthèse d’ADNc non spécifique à un brin amorcé au hasard.
une Fois que vous avez une bibliothèque de séquençage, il est séquencé à une profondeur spécifiée, qui dépend de ce que vous voulez faire avec les données., Ces lectures sont alignées sur le génome ou le transcriptome et sont comptées pour déterminer l’expression différentielle des gènes ou analysées plus avant pour déterminer l’épissage et l’expression isoforme. La plupart des gens séquencent L’ARN en utilisant des méthodes appariées 50-100bp. L’exception est le séquençage des microARN, car cela ne nécessite que le séquençage 36bp à une extrémité dans la plupart des cas.
notre méthode ARN-Seq
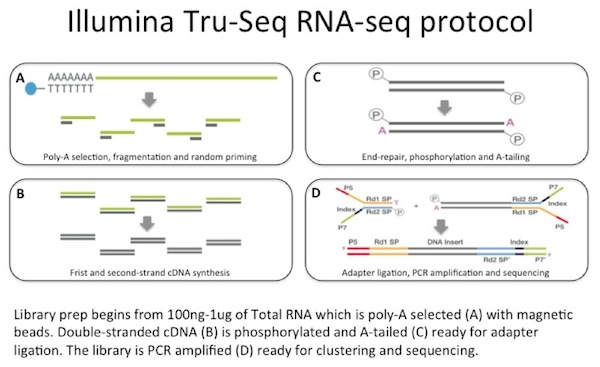
Nous utilisons entre 100 ng et 1 µg d’ARN total comme entrée pour une capture d’ARNm avec des billes magnétiques revêtues d’oligo-dT. L’ARNm est fragmenté, puis une synthèse d’ADNc amorcée au hasard est effectuée., L’ADNc double brin qui en résulte est utilisé comme entrée pour une préparation de bibliothèque Illumina standard qui comprend la réparation finale, la ligature de l’adaptateur et l’amplification PCR pour vous donner une bibliothèque prête pour le séquençage.
Pourquoi s’Embêter Avec un Brin d’Informations?
Il y a eu beaucoup de discussions sur la transcription anti-sensorielle et sa pertinence biologique. Si vous êtes intéressé par une expression génique différentielle simple, les informations sur les brins n’ajouteront pas grand-chose à votre expérience, mais rendront votre protocole plus complexe., Cela dit, Vous pouvez effectuer la méthode la plus largement adoptée sans trop d’effort supplémentaire. Pour ce faire, lors de la synthèse du 2ème brin d’ADNc, utilisez l’uracile pour l’incorporation au lieu de la thymine. Suivez la préparation de la bibliothèque Illumina comme d’habitude, mais après la ligature de l’adaptateur et avant l’amplification par PCR, ajoutez de l’uracile-ADN glycosylase pour dégrader le 2ème brin. Il en résulte que toutes les lectures commencent dans la même orientation afin que vous puissiez déterminer quel brin était transcrit dans votre échantillon.
que Pouvez-vous réellement faire avec RNA-Seq?,
RNA-seq est un outil puissant et polyvalent largement publié ces dernières années. J’ai choisi quelques-uns de mes favoris (certains du travail effectué dans l’installation de base que je gère) pour illustrer ce que vous pouvez faire avec le séquençage de L’ARN.
- Jabbari, et coll. utilisé ARN-seq pour étudier le psoriasis et trouver de nouveaux gènes pour l’analyse fonctionnelle. Ils ont comparé leurs données D’ARN-seq aux études publiées de tableau et ont trouvé 1700 nouveaux candidats. Ceux-ci ont été validés par qPCR, et la comparaison avec des bases de données fonctionnelles pour le psoriasis a confirmé leur rôle dans la pathogenèse.
- Kutter, et coll., utilisé L’ARN-seq dans une étude portant sur la conservation de la liaison de L’ARN polymérase III chez les mammifères pour valider l’expression des gènes occupés par Pol III tel que mesuré par ChIP-seq.
- Mercer, et coll. capture combinée D’ARN-seq et de microréseaux pour identifier et caractériser des transcriptions rares, qui sont normalement indétectables. Les transcriptions ciblées ont augmenté dans l’abondance de lecture séquentielle de 0,21% avant la capture à 80% après la capture., Ils ont trouvé plus de 200 isoformes précédemment non annotées pour près de 50 locus codant des protéines, y compris une nouvelle isoforme alternative de TP53, qui est un gène très bien caractérisé. Cela suggère qu’il y a encore beaucoup de complexité dans le génome et le transcriptome à résoudre.
En résumé, L’ARN-seq est toujours un outil en évolution, mais il est préférable dans la plupart des cas aux puces à puces. Il est plus sensible, plus robuste et peut être plus rentable. Quels projets ARN-seq planifiez-vous maintenant pour votre projet?,
Jabbari et al: le profilage transcriptionnel du Psoriasis à L’aide de L’ARN-seq révèle des gènes précédemment non identifiés exprimés de manière différentielle. Journal of Investigative Dermatology 2011.
Kutter et al: la liaison de Pol III chez six mammifères montre une conservation parmi les isotypes d’acides aminés malgré la divergence entre les gènes de l’ARNt. Nature Genetics 2011.
Levin et al: analyse comparative Complète de volet spécifique de l’ARN méthodes de séquençage. Méthodes Nature 2010.
Mercer et al: le séquençage ciblé de L’ARN révèle la complexité profonde du transcriptome humain. Nature Biotechnology 2012.,
Wang et al: RNA-Seq: un outil révolutionnaire pour la transcriptomique. Nat Rev Genet 2009.
Initialement publié le 25 Mai 2012. Mis à jour et révisé le 16 août 2015.

cela vous a-t-il aidé? Ensuite, veuillez partager avec votre réseau.