– >
RNA-sekvensering (Wang 2009) er raskt bytte genuttrykk microarrays i mange laboratorier. RNA-seq kan du kvantifisere, oppdage og profil RNAs. For denne teknikken, mRNA (og andre RNAs) er først konvertert til cDNA. Den cDNA er deretter brukt som input for neste generasjons sekvensering bibliotek forberedelse. I denne artikkelen vil jeg gi en kort gjennomgang av RNA-seq og introdusere de viktigste metodene som brukes i dag.
Hvorfor Er RNA-Seq «Bedre» Enn Microarrays?,
Det er flere fordeler med RNA-seq har over microarrays:
Med RNA-seq du kan kartlegge mer enn bare differensiert genuttrykk. Selv om det er microarrays tilgjengelig for ekson-nivå og microRNA analyse, de fleste brukere er fortsatt interessert i grunnleggende, sannsynligvis 3′ partisk, differensiert genuttrykk. Med RNA-seq du kan se på kodende og ikke-kodende RNA, ved skjøting og allelspesifikke uttrykk, og kanskje snart i full-lengde cDNA-sekvenser, å eliminere behovet for å utlede eller montere isoforms.,
Microarrays er også partisk, som vi har til å bestemme hva slags innhold til plass på tabellen. Siden RNA-seq ikke bruk prober eller primere, data lider av mye lavere skjevheter (selv om jeg mener ikke å si RNA-seq har ingen).
RNA-seq gir digitale data i form av justert lese-teller, noe som resulterer i et meget bredt dynamisk område, bedre følsomhet for gjenkjenning for sjeldne transkripsjoner.
Det er også veldig kostnad konkurransedyktig for å microarrays, som i dag, mellom 6-30 prøver kan være multiplekset i en enkelt Illumina-sekvensering lane.,
til Slutt, kan du omanalyserer et RNA-seq dataset som mer informasjon om transcriptome blir tilgjengelig. Hvis papiret er publisert viser en interessant skjøte-variant i en lignende system til en du jobber på, så vil du kanskje gå tilbake og se på at skjøting i prøvene, og du vil allerede har data til å gjøre det.
Hvordan Gjør RNA-Seq Arbeid?
Det er mange metoder for å utføre en RNA-seq eksperiment. Faktisk, teknikker utvikler seg så raskt at det kan være vanskelig å bestemme hvilken du skal bruke., Et grunnleggende valg er mellom 1) random-primet cDNA syntese fra dobbel-strandet cDNA eller 2) RNA-ligation metoder (gjennomgått og sammenlignet i Levin 2010). De fleste bruker den første metoden, og deretter trenger for å gjøre en ytterligere valget mellom en strand-spesifikke protokollen, og en som ikke er det. Metoden som brukes mest i min lab er Illumina er TruSeq RNA-seq, som er en tilfeldig-primet cDNA syntese ikke-strand-bestemt protokoll.
Når du har en sekvensering bibliotek, det er sekvensert til en angitt dybde, som er avhengig av hva du ønsker å gjøre med data., Disse leser er justert til genom eller transcriptome og er regnet for å fastsette differensiert genuttrykk eller videre analysert for å fastslå skjøting og isoformen uttrykk. De fleste er RNA-sekvensering ved hjelp av sammenkoblede-end 50-100 bp metoder. Unntaket er microRNA-sekvensering, siden dette krever bare én slutten 36bp sammenheng i de fleste tilfeller.
Våre RNA-Seq Metode
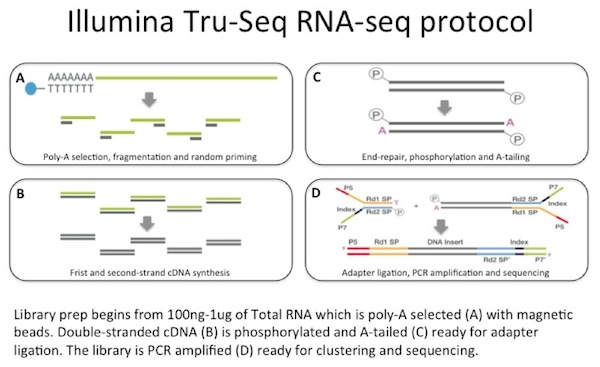
Vi bruke mellom 100 ng til 1 µg total RNA som input til et mRNA ta med oligo-dT belagt magnetisk perler. Den mRNA er fragmentert, og deretter en random-primet cDNA-syntese er utført., Den resulterende dobbel-strand cDNA er brukt som input til en standard Illumina bibliotek prep som inkluderer ende-reparasjon, – adapter ligation og PCR-amplifikasjon for å gi deg et bibliotek klar for sekvensering.
Hvorfor Bry seg Med Strand Informasjon?
Det har vært mye diskusjon om anti-følelse transkripsjon og dets biologiske relevans. Hvis du er interessert i enkle differensial genuttrykk, deretter strand informasjon vil ikke legge mye til eksperimentet, men vil gjøre dine protokollen mer komplekse., Etter å ha sagt at, du kan utføre de mest allment vedtatt metode uten for mye ekstra innsats. For å gjøre dette, under 2. strand cDNA syntese, bruk uracil for inkorporering i stedet for innhold av tymin. Følg Illumina bibliotek prep som normalt, men etter adapter ligation og før PCR-amplifikasjon legge til uracil-DNA glycosylase å forringe 2ndstrand. Dette resulterer i at alle leser starter i samme retning, slik at du kan finne ut hvilken strand ble transkribert i ditt eksempel.
Hva Kan Du Egentlig Gjøre Med RNA-Seq?,
RNA-seq er et kraftig og fleksibelt verktøy som er publisert mye i løpet av de siste årene. Jeg har plukket ut et par av mine favoritter (noen fra arbeid som utføres i core facility jeg klarer) å illustrere hva du kan gjøre med RNA-sekvensering.
- Jabbari, et al. brukes RNA-seq å undersøke psoriasis og finne nye gener for funksjonell analyse. De sammenlignet sin RNA-seq data publisert utvalg studier, og fant 1700 nye kandidater. Disse ble validert av qPCR, og sammenligningen til funksjonelle databaser for psoriasis støttet deres rolle i patogenesen.
- Kutter, et al., brukes RNA-seq i en studie ser på bevaring av RNA Polymerase III bindende i pattedyr som brukes til å validere uttrykket av gener som er okkupert av Pol III som analysert med ChIP-seq.
- Mercer, et al. kombinert RNA-seq og microarray-basert ta bilde for å identifisere og karakterisere sjeldne transkripsjoner, som normalt er umulig å oppdage. Målrettet transkripsjoner økt i rekkefølge les overflod fra 0.21% pre-fange 80% innlegget fange., De fant mer enn 200 tidligere unannotated isoforms for nesten 50 protein-kodende loci, inkludert et nytt alternativ isoformen av TP53, som er et svært godt preget genet. Dette tyder på at det fortsatt er mye kompleksitet i genomet og transcriptome til å være løst.
I sammendraget, RNA-seq er fortsatt en samling verktøy, men er å foretrekke i de fleste tilfeller å microarrays. Det er mer følsom, mer robust, og kan være mer kostnadseffektivt. Hva RNA-seq prosjekter er du nå planlegger for ditt prosjekt?,
Jabbari et al: Transcriptional Profilering av Psoriasis ved Hjelp av RNA-seq Avslører Tidligere Uidentifiserte Differentially Uttrykte Gener. Journal of Investigative Dermatologi 2011.
Kutter et al: Pol III bindende i seks pattedyr viser bevaring blant aminosyre isotypes til tross for ulikhetene mellom tRNA-gener. Arten Genetikk 2011.
Levin et al: Omfattende komparativ analyse av strand-spesifikke RNA-sekvensering metoder. Arten Metoder 2010.
Mercer et al: Målrettet RNA-sekvensering avslører dype kompleksiteten i den menneskelige transcriptome. Arten Bioteknologi 2012.,
Wang et al: RNA-Seq: et revolusjonerende verktøy for transcriptomics. Nat Rev Genet 2009.
Opprinnelig publisert på Mai 25, 2012. Oppdatert og revidert 16. August 2015.

Har dette hjulpet deg? Så vær så snill å dele med nettverket.