het rangschikken van RNA (Wang 2009) vervangt snel genexpressie microarrays in vele laboratoria. RNA-seq laat u RNAs kwantificeren, ontdekken en profileren. Voor deze techniek, worden mRNA (en andere RNAs) eerst omgezet in cDNA. CDNA wordt dan gebruikt als input voor een volgende generatie het rangschikken bibliotheekvoorbereiding. In dit artikel, zal ik een kort overzicht van RNA-seq geven en de belangrijkste methodes introduceren die vandaag worden gebruikt.
Waarom is RNA-Seq “beter” dan Microarrays?,
Er zijn verscheidene voordelen RNA-seq heeft over microarrays:
met RNA-seq kunt u meer dan enkel differentiële genuitdrukking ondervragen. Hoewel er microarrays beschikbaar voor exon-niveau en microRNA analyse zijn, zijn de meeste gebruikers nog geinteresseerd in basis, waarschijnlijk 3′ bevooroordeelde, differentiële genuitdrukking. Met RNA-seq kunt u codage en niet-codage RNA bekijken, bij het verbinden en allelen specifieke uitdrukking, en misschien spoedig bij volledige cDNA opeenvolgingen, eliminerend de behoefte om isovormen af te leiden of te assembleren.,
Microarrays zijn ook bevooroordeeld, omdat we moeten beslissen welke inhoud we op de array plaatsen. Aangezien RNA-seq geen sondes of inleidingen gebruikt, lijden de gegevens aan veel lagere vooroordelen (hoewel ik niet bedoel te zeggen RNA-seq heeft geen).
RNA-seq verstrekt digitale gegevens in de vorm van uitgelijnde read-tellingen, resulterend in een zeer breed dynamisch bereik, die de gevoeligheid van opsporing voor zeldzame transcripten verbeteren.
Het is ook zeer kosten-concurrerend aan microarrays, aangezien vandaag, tussen 6-30 steekproeven in één enkele Illumina het rangschikken baan kunnen worden gemultiplext.,
ten slotte kunt u een RNA-seq-dataset opnieuw analyseren naarmate meer informatie over het transcriptoom beschikbaar komt. Als een paper wordt gepubliceerd met een interessante splice-variant in een vergelijkbaar systeem als het systeem waar je aan werkt, dan zou je terug willen gaan en kijken naar die splicing in je samples; en je zou al de gegevens hebben om dat te doen.
Hoe werkt RNA-Seq?
Er zijn vele methoden voor het uitvoeren van een RNA-seq experiment. In feite evolueren de technieken zo snel dat het moeilijk kan zijn om te beslissen welke te gebruiken., Een basiskeuze is tussen 1) random-primed cDNA synthese van double-stranded cDNA of 2) RNA-ligation methodes (beoordeeld en vergeleken in Levin 2010). De meeste mensen gebruiken de eerste methode en moeten dan een verdere keuze maken tussen een streng-specifiek protocol en een protocol dat dat niet is. De methode die het meest in mijn laboratorium wordt gebruikt is TruSeq RNA-seq van Illumina, die een random-primed cDNA synthese non-Bundle-specific protocol is.
zodra u een sequentiebibliotheek hebt, wordt deze gesequenced tot een bepaalde diepte, die afhankelijk is van wat u met de gegevens wilt doen., Deze leest worden afgestemd op het genoom of transcriptome en geteld om differentiële genuitdrukking te bepalen of verder geanalyseerd om het verbinden en isovormuitdrukking te bepalen. De meeste mensen rangschikken RNA gebruikend in paren gerangschikt-eind 50-100bp methodes. De uitzondering is microRNA het rangschikken, aangezien dit slechts het single-end 36bp rangschikken in de meeste gevallen vereist.
onze RNA-Seq-methode
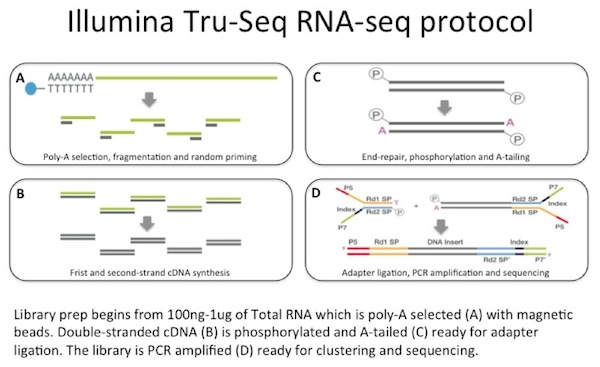
we gebruiken 100 ng tot 1 µg totaal RNA als input voor een mRNA-opname met oligo-dT gecoate magnetische parels. MRNA is gefragmenteerd, en dan wordt een random-primed cDNA synthese uitgevoerd., De resulterende dubbel-bundel cDNA wordt gebruikt als input aan een standaard Illumina-bibliotheekvoorbereiding die eind-reparatie, adapter ligation en PCR-versterking omvat om u een bibliotheek klaar te geven voor het rangschikken.
Er is veel discussie geweest over anti-sense transcriptie en de biologische relevantie ervan. Als u in eenvoudige differentiële genuitdrukking geinteresseerd bent, dan zal de bundelinformatie niet veel aan uw experiment toevoegen, maar zal uw protocol complexer maken., Dat gezegd hebbende, kunt u de meest gebruikte methode uitvoeren zonder al te veel extra inspanning. Om dit te doen, tijdens 2de bundel cDNA synthese, gebruik uracil voor incorporatie in plaats van thymine. Volg de Illumina bibliotheek prep zoals normaal, maar na adapter ligation en vóór PCR versterking voeg uracil-DNA glycosylase toe om 2ndstrand te degraderen. Dit resulteert in alle leest die in dezelfde oriëntatie beginnen zodat u kunt bepalen welke streng in uw steekproef werd getranscribeerd.
wat kun je eigenlijk doen met RNA-Seq?,
RNA-seq is een krachtig en veelzijdig hulpmiddel dat de laatste jaren op grote schaal is gepubliceerd. Ik heb een paar van mijn favorieten (sommige van het werk uitgevoerd in de kern faciliteit ik beheren) om te illustreren wat je kunt doen met RNA-sequencing.
- Jabbari, et al. gebruikt RNA-seq om psoriasis te onderzoeken en nieuwe genen voor functionele analyse te vinden. Zij vergeleken hun RNA-seq gegevens aan gepubliceerde array studies en vonden 1700 nieuwe kandidaten. Deze werden gevalideerd door qPCR, en vergelijking met functionele databases voor psoriasis ondersteunde hun rol in pathogenese.
- Kutter, et al., gebruikte RNA-seq in een studie die het behoud van de polymerase III van RNA bekijken band in zoogdieren om uitdrukking van genen te valideren die door Pol III worden bezet zoals geanalyseerd door Spaander-seq.
- Mercer, et al. gecombineerde vangst van RNA-seq en microarray-gebaseerd om zeldzame transcripten te identificeren en te karakteriseren, die normaal niet op te sporen zijn. De gerichte afschriften stegen in opeenvolging gelezen overvloed van 0,21% pre-capture aan 80% post capture., Zij vonden meer dan 200 eerder niet-geannoteerde isovormen voor bijna 50 eiwit-coderende loci, met inbegrip van een nieuwe alternatieve isovorm van TP53, die een zeer goed gekenmerkt gen is. Dit stelt voor dat er nog veel complexiteit in het genoom en transcriptome moet worden opgelost.
samengevat is RNA-seq nog steeds een evoluerend hulpmiddel, maar is in de meeste gevallen beter dan microarrays. Het is gevoeliger, robuuster en kan kosteneffectiever zijn. Welke RNA-seq projecten plant u nu voor uw project?,
Jabbari et al: transcriptionele profilering van Psoriasis met behulp van RNA-seq onthult eerder niet-geïdentificeerde differentieel tot expressie gebrachte genen. Journal of Investigative Dermatology 2011.
Kutter et al: Pol III-binding bij zes zoogdieren vertoont behoud bij aminozuurisotypen ondanks divergentie tussen tRNA-genen. Nature Genetics 2011.
Levin et al: Comprehensive comparative analysis of strand-specific RNA sequencing methods. Natuurmethoden 2010.
Mercer et al: gerichte RNA-sequencing onthult de diepe complexiteit van het menselijke transcriptoom. Natuur Biotechnologie 2012.,Wang et al: RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet 2009.
oorspronkelijk gepubliceerd op 25 mei 2012. Bijgewerkt en herzien op 16 augustus 2015.

heeft dit u geholpen? Dan kunt u delen met uw netwerk.